Hyperactive pG-MNase CUT&RUN Assay Kit for Illumina
快速高效靶向切割目的片段,文库构建优质且真实。

操作体验好:实验流程简单,包含DNA提取模块,告别酚氯仿抽提。
产品性能好:转录因子、辅因子成功率高,产出更稳定,结果更真实。
结果更准确:含有Spike in DNA组分,校正组间差异。

Hyperactive pG-MNase CUT&RUN Assay Kit for Illumina是针对Illumina高通量测序平台定向开发的用于研究蛋白质-DNA相互作用的试剂盒。CUT&RUN(Cleavage Under Target and Release Using Nuclease)技术是一种研究蛋白质-DNA相互作用的新方法,使用Protein G融合的MNase核酸酶,在抗体引导下精准靶向目的蛋白,并在目的位点附近进行DNA的片段化切割。本试剂盒优化了实验反应体系和建库流程,与传统的ChIP-Seq相比,具有实验成功率高、抗体兼容性强、实验周期短、操作简单等优势,尤其适用于早期胚胎发育、干细胞、肿瘤以及表观遗传学等研究领域。试剂盒中所有试剂都经过严格的质量控制和功能验证,较大程度上保证了文库构建的稳定性和重复性。

1. 优质的文库产出
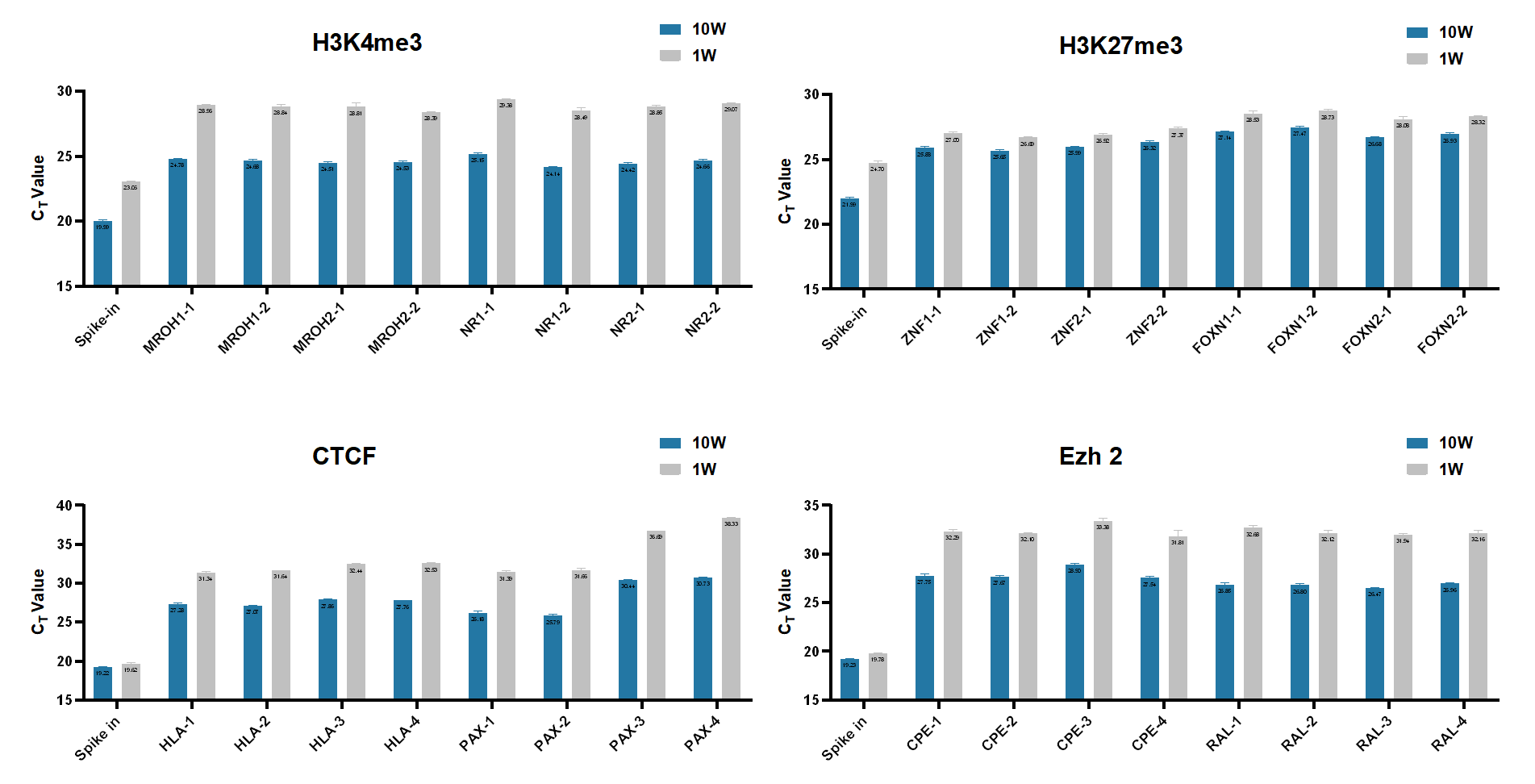
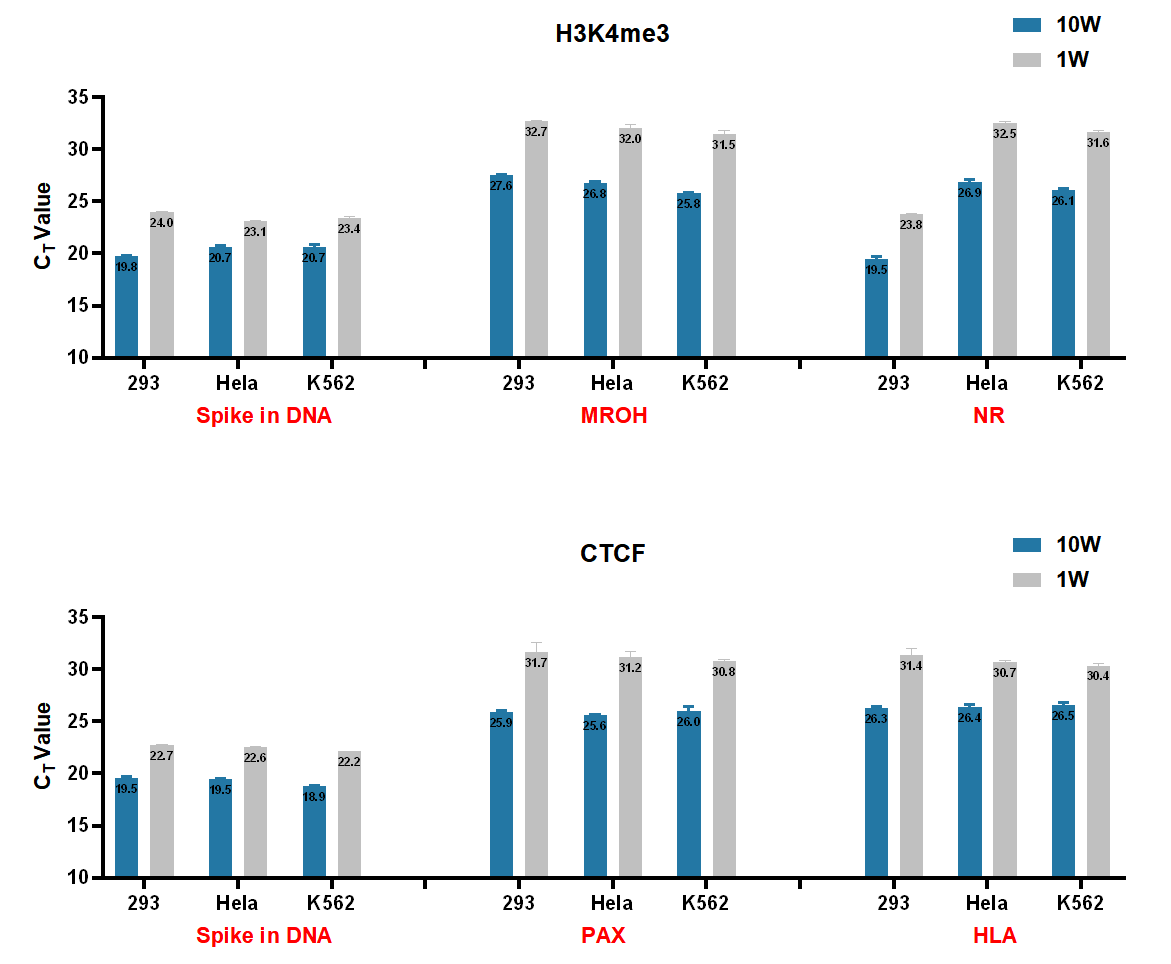
以100,000个K562细胞为样本,参照HD102实验流程对不同丰度靶蛋白进行CUT&RUN实验和文库构建。在不同组蛋白和转录因子体系中,HD102文库产出稳定,文库峰型呈现典型的Ladder状分布,HD102精准靶向和高效切割的特性保证了优质的文库产出。
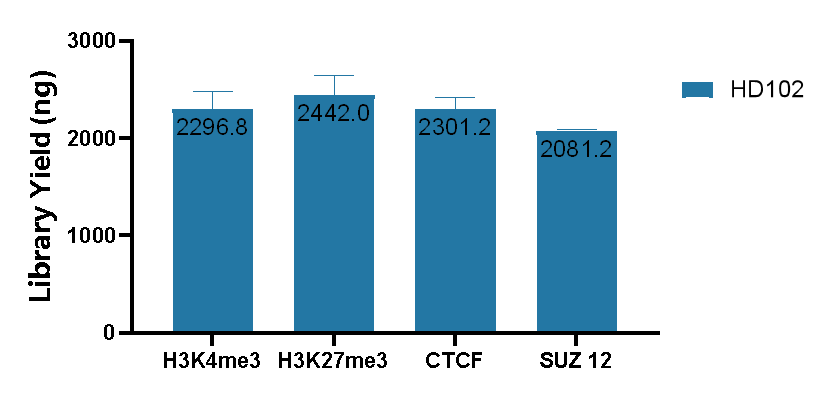
图1 HD102文库产出

图2 HD102文库峰型
2. 高质量的下机数据
以100,000个K562细胞为样本,参照HD102实验流程对不同丰度靶蛋白进行CUT&RUN实验和文库构建。通过HD102获得的下机数据TSS富集以及IGV视图信噪比高,对目的基因位点有明显富集。

图3 HD102 TSS富集图
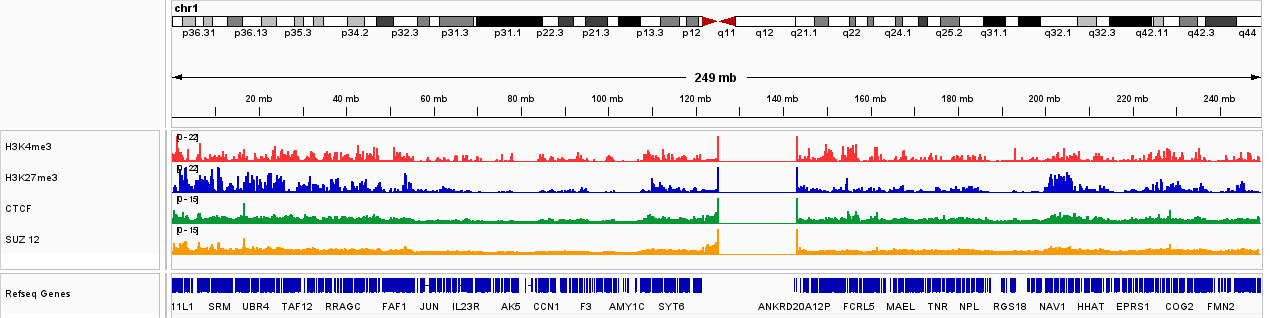
图4 HD102 Chr1 IGV全局视图
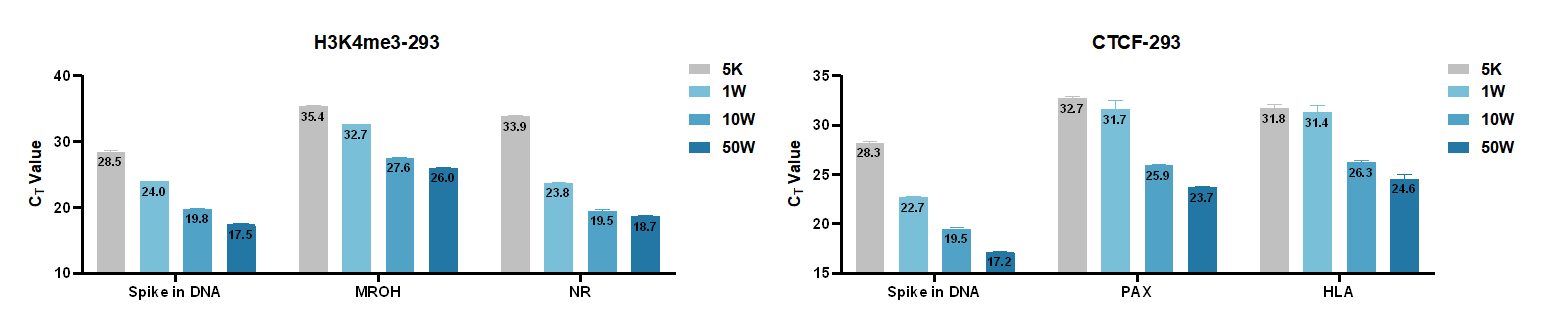
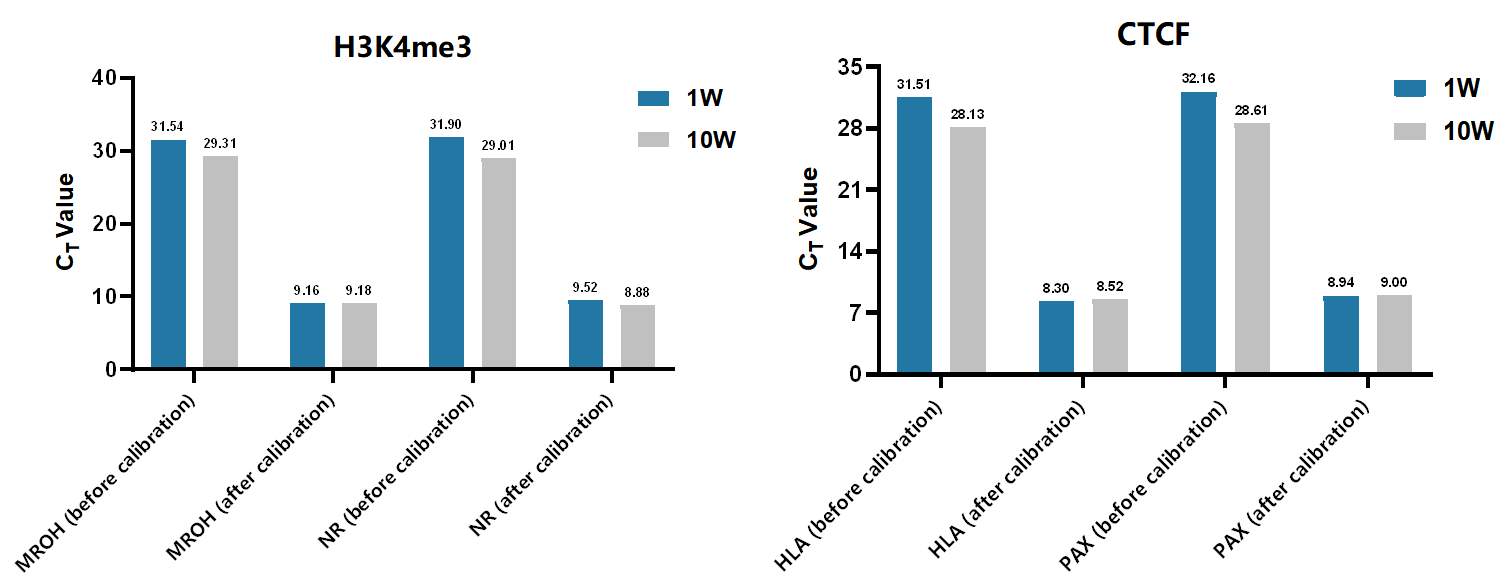
3. Spike in DNA校正组间差异
以5,000、10,000、100,000个293细胞为样本,参照HD102实验流程对组蛋白H3K4me3进行CUT&RUN实验和文库构建,并按照说明书推荐的Spike in DNA投入量进行校正。使用HD102建库获得的热图和Plotfile在校准前由于高低细胞投入量获得的数据量的差异,导致富集效果存在显著差异,通过Spike in DNA进行均一化校准之后,可以真实反应不同细胞投入量下的富集效果。
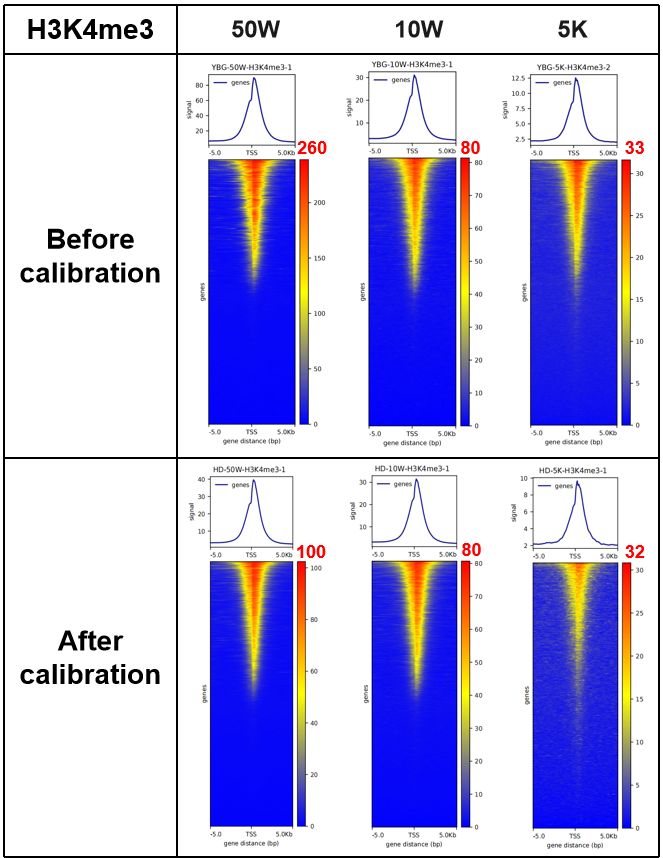
图5 HD102 Spike in DNA校正前后TSS富集比较
4. 多组学比较
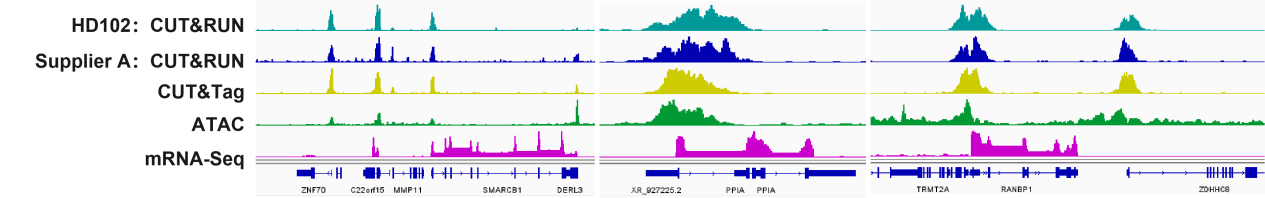
以100,000个293细胞为样本进行CUT&RUN、CUT&Tag、ATAC和mRNA-seq多组学比较分析。IGV视图显示HD102在关键位点上的富集情况与ATAC和CUT&Tag技术一致,且在mRNA-seq中可以找到相应基因位点,实验结果真实可靠。

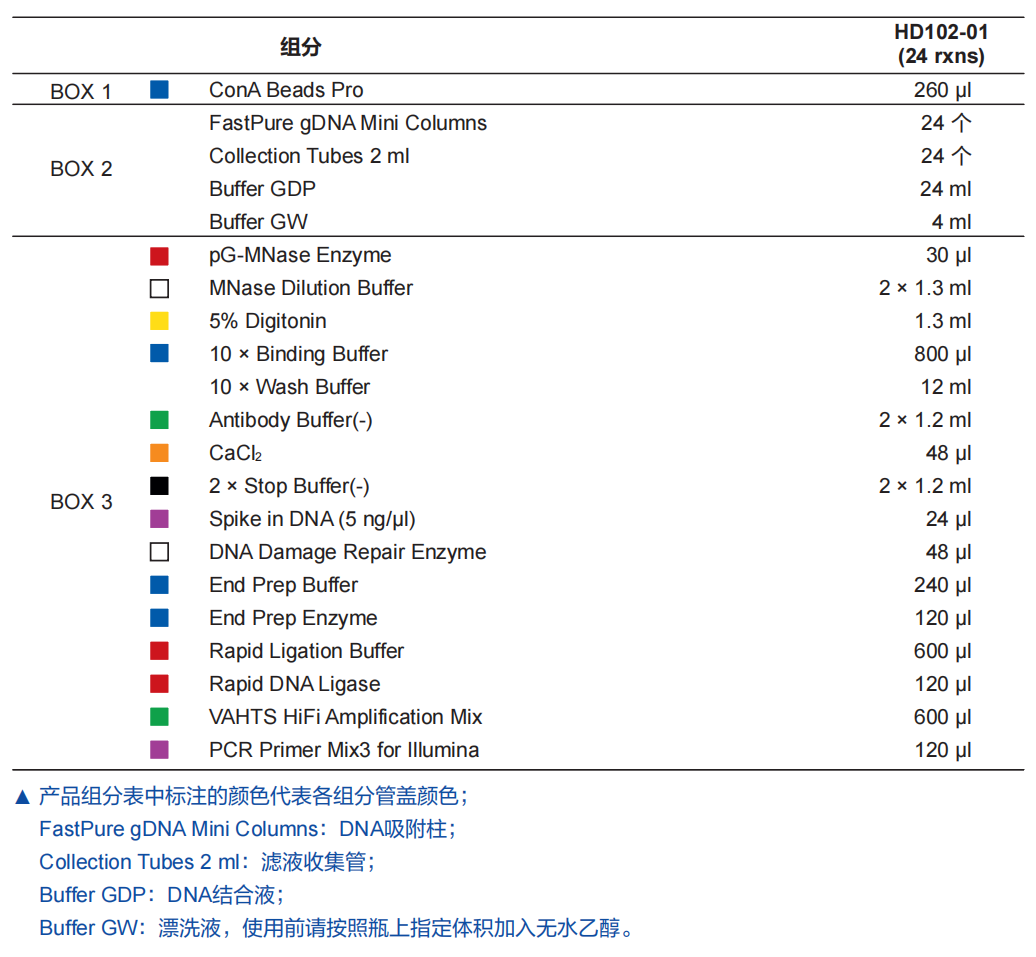

BOX1:2 ~ 8 ℃保存,根据不同目的地调整运输方式;
BOX2:15 ~ 25 ℃保存,常温运输;
BOX3:-30 ~ -15 ℃保存,≤0℃运输。