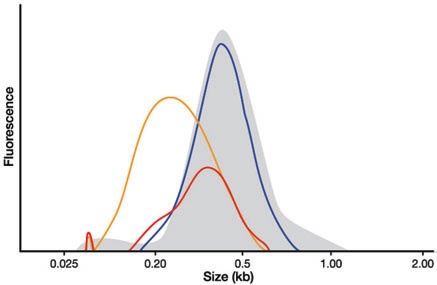
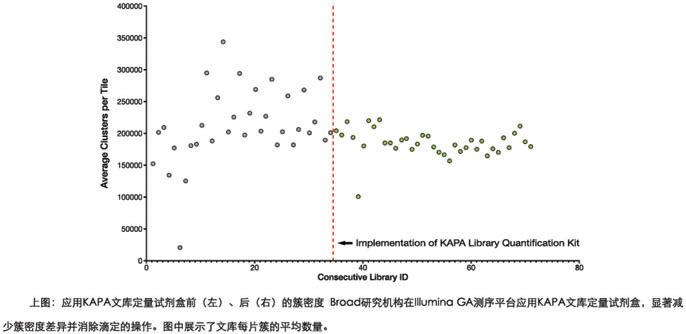
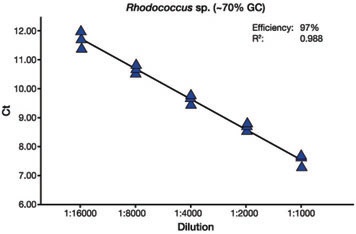
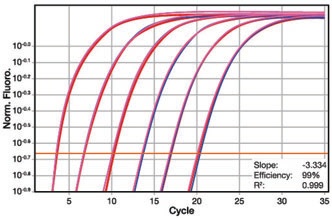
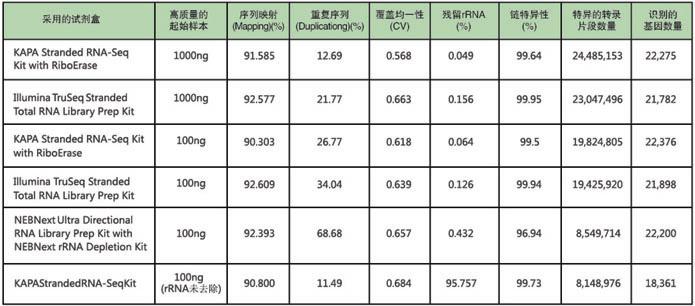
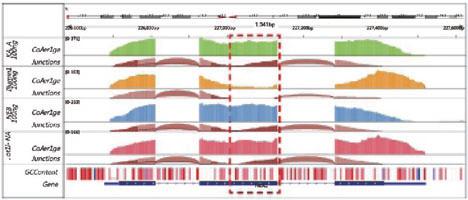
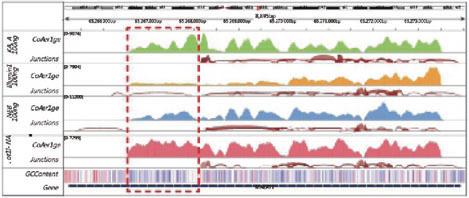
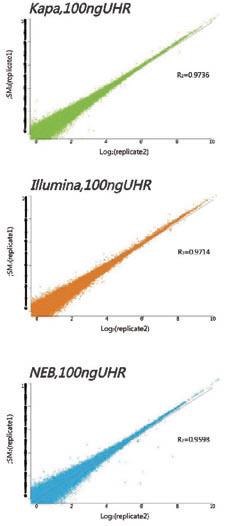
KAPA KK2631 热启动HiFi高保真酶 HiFi HotStart ReadyMix PCR Kit
KK2631 KAPA热启动HiFi高保真酶预混液50mL
英文名称:KAPA HiFi HotStart ReadyMix Kit (50mL)
产地/品牌:kapa
规 格:50mL/2000*50μL rxn/4000*25μL rxn 2000
货 号:KK2631
上海金畔生物代理Kapa biosystems品牌产品,我们将竭诚为您服务,欢迎访问Kapa biosystems官网或者咨询我们获取更多相关Kapa biosystems品牌产品信息。
Product Description 产品描述
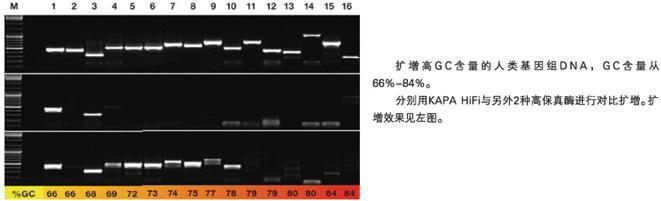
KAPA HiFi HotStart DNA Polymerase is a novel B-family DNA polymerase, engineered to have increased affinity for DNA, without the need for accessory proteins or DNA binding domains. The intrinsic high processivity of the enzyme results in significant improvement in yield, speed and sensitivity when compared with wild-type B-family DNA polymerases. In addition, the ability to amplify long targets, as well as GC- and AT-rich targets, is significantly improved. The enzyme is combined with a proprietary antibody that inactivates the enzyme until the first denaturation step. This prevents nonspecific amplification during reaction setup, increases sensitivity, and improves reaction efficiency.
KAPA HiFi HotStart DNA 聚合酶是一种新型 B 家族 DNA 聚合酶,经过精心设计,可增强对 DNA 的亲和力,无需辅助蛋白或 DNA 结合结构域。 与野生型 B 家族 DNA 聚合酶相比,该酶固有的高持续合成能力导致产量、速度和灵敏度显着提高。 此外,扩增长靶标以及富含 GC 和 AT 的靶标的能力显着提高。 该酶与一种专有抗体相结合,使酶失活直到第一个变性步骤。 这可以防止反应设置期间的非特异性扩增,提高灵敏度并提高反应效率。
In the ReadyMix PCR Kit, KAPA HiFi HotStart DNA Polymerase is supplied in a convenient 2X ReadyMix format, containing all reaction components except primers and template. The ReadyMix contains KAPA HiFi HotStart DNA Polymerase (0.5 U per 25 μL reaction) in a proprietary reaction buffer containing dNTPs (0.3 mM of each dNTP at 1X), MgCl2 (2.5 mM at 1X) and stabilizers.
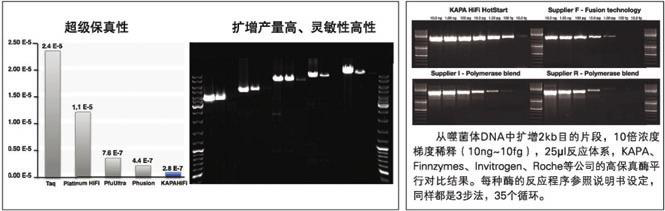
KAPA HiFi HotStart ReadyMix is designed for routine, high-fidelity PCR of a wide range of targets and fragment sizes. It offers error rates approximately 100 times lower than wild-type Taq DNA polymerase, and higher success rates and yields than achievable with wild-type B-family (proofreading) DNA polymerases. In addition, KAPA HiFi HotStart requires significantly shorter reaction times than wild-type B-family DNA polymerases.
KAPA HiFi HotStart DNA Polymerase has 5’→3’ polymerase and 3’→5’ exonuclease (proofreading) activity, but no 5’→3’ exonuclease activity. The strong 3’→5’ exonuclease activity results in superior accuracy during DNA amplification, lending to KAPA HiFi HotStart DNA Polymerase the lowest published error rate of all B-family DNA polymerases (1 error per 3.6 x 106 nucleotides incorporated). This fidelity is approximately 100 times higher than that of wild-type Taq DNA polymerase, and up to 10 times higher than that of other B-family DNA polymerases and polymerase blends.
DNA fragments generated with KAPA HiFi HotStart ReadyMix may be used for routine downstream analysis and applications, including restriction enzyme digestion, cloning and sequencing. PCR products generated with KAPA HiFi HotStart ReadyMix are blunt-ended, but may be 3’-dA-tailed for cloning into TA cloning vectors (see Important Parameters: TA cloning).
Product Applications 产品应用
The KAPA HiFi HotStart PCR Kit is ideally suited for:
• PCR for conventional sequencing (direct sequencing or sequencing of cloned PCR products)
• Next-generation sequencing library amplification (when used in conjunction with the KAPA Library Amplification protocol)
• Amplification of DNA fragments for cloning and protein expression or genomic characterization
• Site-directed mutagenesis.
For more information on these and other high-fidelity PCR applications, please refer to the KAPA HiFi Application Notes on Site-Directed Mutagenesis, Routine High-Fidelity PCR, and High-Fidelity GC-rich PCR available from www.kapabiosystems.com.
KAPA HiFi HotStart PCR 试剂盒非常适合:
• 用于常规测序的 PCR(直接测序或克隆 PCR 产物的测序)
• 下一代测序文库扩增(与 KAPA 文库扩增方案结合使用时)
• 用于克隆和蛋白质表达或基因组表征的 DNA 片段扩增
• 定点诱变。
有关这些和其他高保真 PCR 应用的更多信息,请参阅 www.kapabiosystems.com 上关于定点诱变、常规高保真 PCR 和高保真 GC-rich PCR 的 KAPA HiFi 应用说明。
Standard PCR Protocol
IMPORTANT! The KAPA HiFi HotStart ReadyMix contains an engineered B-family (proofreading) DNA polymerase and uniquely-formulated buffers, and requires specialized reaction conditions. If these conditions are not adhered to, reaction failure is likely. Refer to Important Parameters for more information.
Step 1: Prepare the PCR master mix
• KAPA HiFi HotStart reactions MUST be set up on ice since the high proofreading activity of the enzyme will result in rapid primer degradation at room temperature.
• Ensure that all reagents are properly thawed and mixed.
• Prepare a PCR master mix containing the appropriate volume of all reaction components common to all or a subset of reactions to be performed.
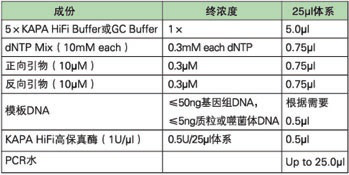
• Calculate the required volumes of each component based on the following table:
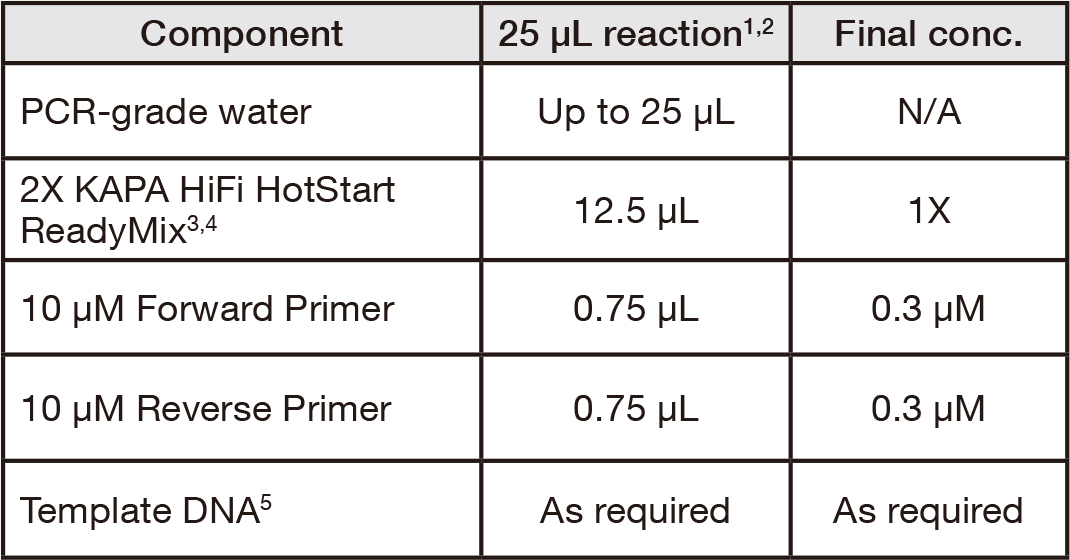
1 Reaction volumes may be adjusted between 10–50 μL. For volumes other than 25 μL, scale reagents proportionally. Reaction volumes > 50 μL are not recommended.
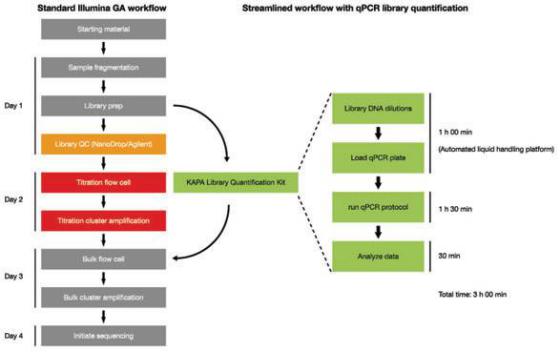
2 When using for next-generation sequencing library amplification, please use protocol provided with the KAPA Library Amplification Kit.
3 KAPA HiFi HotStart ReadyMix contains 2.5 mM MgCl2 (1X). Additional MgCl2 may be added separately, but is unlikely to be required.
4 KAPA HiFi HotStart ReadyMix contains 0.3 mM of each dNTP (1X), and 0.5 U of KAPA HiFi HotStart DNA Polymerase (per 25 μL reaction) in a proprietary reaction buffer.
5 Use < 100 ng genomic DNA (10–100 ng) and <1 ng less complex DNA (0.1–1 ng) per 25 μL reaction as first approach.
Step 2: Set up individual reactions
• Transfer the appropriate volumes of PCR master mix, template and primer to individual PCR tubes or wells of a PCR plate.
• Cap or seal individual reactions, mix and centrifuge briefly.
Step 3: Run the PCR
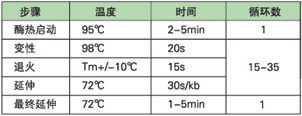
• Perform PCR with the following cycling protocol1:
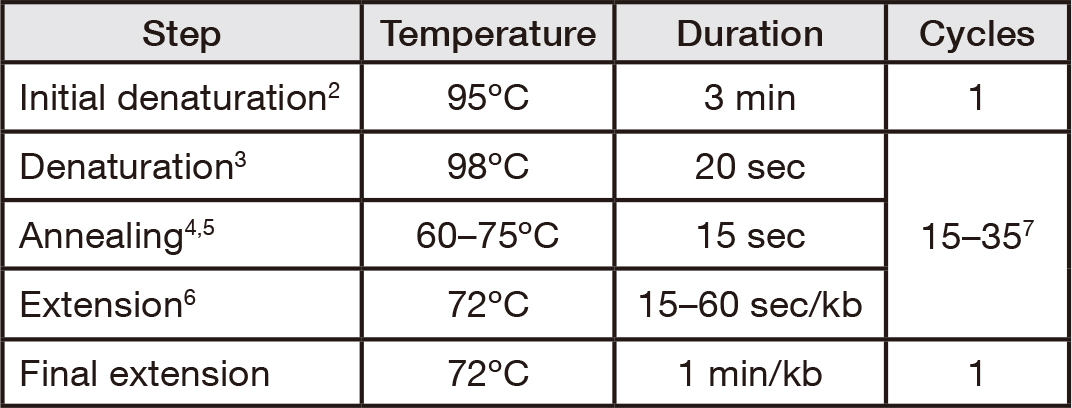
1 When using for next-generation sequencing library amplification, please use protocol provided with the KAPA Library Amplification Kit.
2 Initial denaturation for 3 min at 95 °C is sufficient for most applications. Use 5 min at 95 °C for GC-rich targets (> 70% GC content).
3 KAPA HiFi HotStart ReadyMix has a higher salt concentration than conventional PCR ready-mixes, which affects DNA melting. To ensure that complex and GC rich targets are completely denatured, use a temperature of 98 °C for denaturation during cycling.
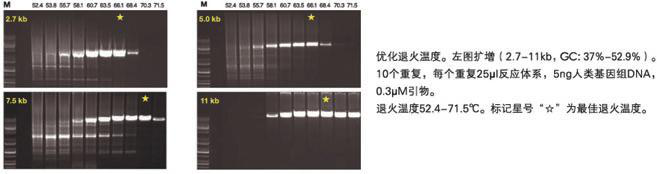
4 In addition to DNA melting, high salt also affects primer annealing. The optimal annealing temperature for a specific primer set is likely to be different (higher) than when used in a conventional PCR ready-mix. An annealing temperature gradient PCR is recommended to determine the optimal annealing temperature with the KAPA HiFi HotStart ReadyMix. If gradient PCR is not feasible, anneal at 65 °C as a first approach.
5 Two-step cycling protocols with a combined annealing/extension temperature in the range of 68–75 °C and a combined annealing/extension time of 30 sec/kb may be used.
6 Use 15 sec extension per cycle for targets ≤ 1 kb, and 30–60 sec/kb for longer fragments, or to improve yields.
7 For highest fidelity, use ≤ 25 cycles. In cases where very low template concentrations or low reaction efficiency results in low yields, 30–35 cycles may be performed to produce sufficient product for downstream applications.
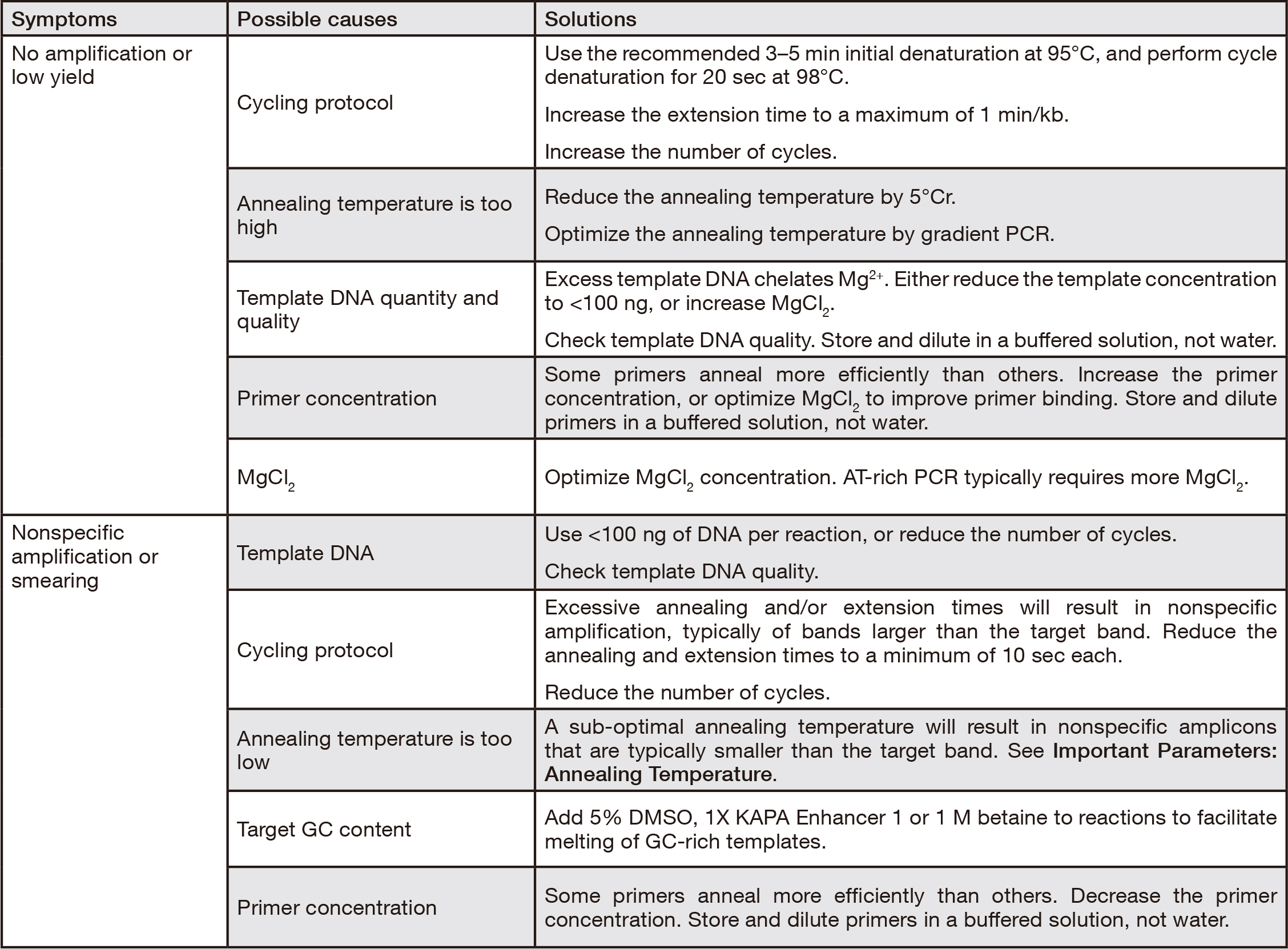
Troubleshooting
| Product Line |
KAPA Code |
Product Chinese Name |
Product Description |
Spec |
Rxn |
| HiFi & Lib Amp |
KK2101 |
KK2101 KAPA HiFi高保真酶+dNTPs (100反应) |
KAPA HiFi+dNTPs (100rxn) |
100*50μL rxn/200*25μL rxn |
100 |
| HiFi & Lib Amp |
KK2102 |
KK2102 KAPA HiFi高保真酶+dNTPs (250反应) |
KAPA HiFi+dNTPs (250rxn) |
250*50μL rxn/500*25μL rxn |
250 |
| HiFi & Lib Amp |
KK2501 |
KK2501 KAPA 热启动HiFi高保真酶+dNTPs (100反应) |
KAPA HiFi HotStart+dNTPs (100rxn) |
100*50μL rxn/200*25μL rxn |
100 |
| HiFi & Lib Amp |
KK2502 |
KK2502 KAPA 热启动HiFi高保真酶+dNTPs (250反应) |
KAPA HiFi HotStart+dNTPs (250rxn) |
250*50μL rxn/500*25μL rxn |
250 |
| HiFi & Lib Amp |
KK2601 |
KK2601 KAPA 热启动HiFi高保真酶预混液 (1.25mL) |
KAPA HiFi HotStart ReadyMix Kit (1.25mL) |
1.25mL/50*50μL rxn/100*25μL rxn |
50 |
| HiFi & Lib Amp |
KK2602 |
KK2602 KAPA 热启动HiFi高保真酶预混液 (6.25mL) |
KAPA HiFi HotStart ReadyMix Kit (6.25mL) |
6.25mL/250*50μL rxn/500*25μL rxn |
250 |
| HiFi & Lib Amp |
KK2631 |
KK2631 KAPA 热启动HiFi高保真酶预混液 (50mL) |
KAPA HiFi HotStart ReadyMix Kit (50mL) |
50mL/2000*50μL rxn/4000*25μL rxn |
2000 |
| HiFi & Lib Amp |
KK2625 |
KK2625 KAPA 热启动HiFi高保真酶预混液 (50mL) v2 |
KAPA HiFi HotStart ReadyMix Kit (50mL) v2 |
50mL/2000*50μL rxn/4000*25μL rxn |
2000 |
| HiFi & Lib Amp |
KK2801 |
KK2801 KAPA Uracil+热启动HiFi高保真酶预混液 (1.25mL) |
KAPA HiFi HotStart Uracil+ ReadyMix Kit (1.25mL) |
1.25mL/50*50μL rxn/100*25μL rxn |
50 |
| HiFi & Lib Amp |
KK2802 |
KK2802 KAPA Uracil+热启动HiFi高保真酶预混液 (6.25mL) |
KAPA HiFi HotStart Uracil+ ReadyMix Kit (6.25mL) |
6.25mL/250*50μL rxn/500*25μL rxn |
250 |