Hyperactive® pG-MNase for CUT&RUN

操作简单:利用酶切代替超声破碎,仅需1天即可实现从细胞到二代测序文库的转化
高活性:DNA片段化活性较高
细胞起始量低:可兼容低至50个细胞
纯度高:蛋白纯度高、核酸残留低

应用于CUT&RUN技术
CUT&RUN(cleavage under targets and release using nuclease)是替换传统的ChIP-Seq用以研究蛋白质-基因组互作的新技术,与后者相比,它具有显著优势,如:信噪比高,可重复性好,实验周期短(1天实现从细胞到文库构建),细胞投入量低等,因此,该方法适用于表观遗传学、肿瘤、干细胞等研究领域。
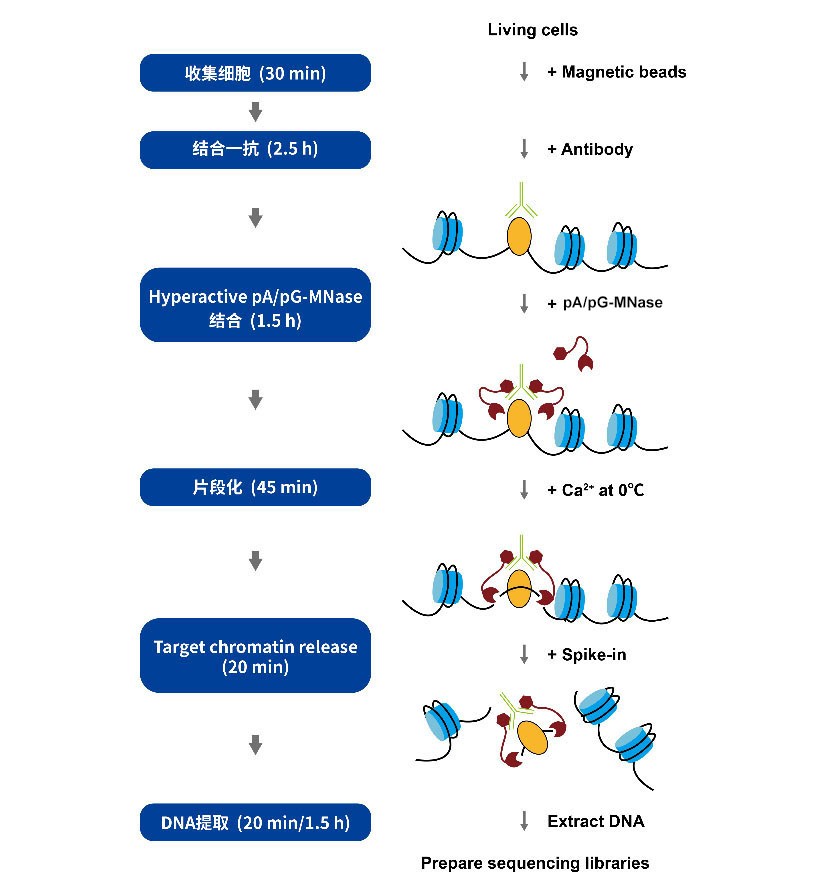
CUT&RUN技术的实验流程简单,主要包括:
1.收集细胞并将细胞与连有伴刀豆蛋白A的磁珠进行结合;
2.孵育一抗;
3.孵育pA/pG-MNase;
4.激活pA/pG-MNase进行片段化;
5.DNA提取;
6.文库构建。
活性检测
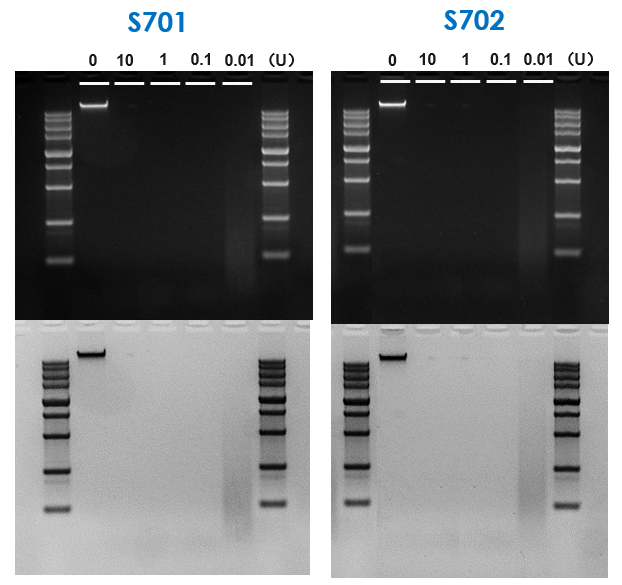
如图所示,针对300 ng人源基因组DNA,梯度加入不同量的S701或S702,于37℃进行酶切反应10 min。只需要0.1U 新型融合核酸酶即可实现300 ng人源基因组DNA片段化,表明该融合酶活性高。

Hyperactive® pG-MNase for CUT&RUN是将Protein G与微球菌核酸酶(Micrococcal nuclease,MNase)进行融合,形成同时具备Protein G与MNase双重活性的新型融合酶,专门适用于蛋白质-基因组互作研究的CUT&RUN技术。CUT&RUN技术简化了传统ChIP-seq的操作流程,仅需1 – 1.5天即可实现从细胞到二代测序文库构建,且细胞投入量低,信噪比高,可重复性好,广泛应用于早期胚胎发育、干细胞、肿瘤以及表观遗传学等研究领域。

|
组分 |
S702-01(200 U) |
S702-02(400 U) |
|
Hyperactive® pG-MNase (10U/μl) |
20 μl |
40 μl |
|
MNase Dilution Buffer |
200 μl |
400 μl |

于-30 ~ -15℃保存。运输条件:≤ 0℃。
