产品描述
Illumina平台的KAPA文库定量试剂盒提供了基于qPCR的Illumina文库绝对定量所需的全部试剂,这些文库两端带有P5和P7流动槽寡核苷酸序列。试剂盒包含:
● 文库定量DNA标准品1-6(线性、452 bp模板的10倍稀释系列)
● Library Quantification Primer Premix 文库定量引物预混液(10x),含有以下引物:
引物1:5′-AAT GAT ACG GCG ACC ACC GA-3′
引物2:5′-CAA GCA GAA GAC GGC ATA CGA-3′
● KAPA SYBR® FASTqPCR预混液(2x),含有各种被动参比染料(表1)。
表1. 推荐用于KAPA SYBR FAST通用qPCR预混液的ROX浓度

文库定量的方法为:通过使用KAPA SYBR FASTqPCR预混液和靶向Illumina P5和P7流动槽寡核苷酸序列的引物,使用qPCR扩增一组六份预稀释的DNA标准品和稀释的文库样本,进行文库定量。将每份DNA标准品的平均Cq值取log10(以pM表示浓度)作图,以生成标准曲线。然后使用绝对定量,通过标准曲线计算稀释的文库样本的浓度。
KAPA文库定量试剂盒经过严格测试,以确保最小的批次间差异。该试剂盒包含新型KAPA SYBR FAST DNA聚合酶,一种通过定向进化工程优化,适用于SYBR Green I的高性能qPCR酶。工程优化的聚合酶能够以相似的效率扩增多种DNA片段,因此能够使用通用的标准品可靠地定量平均片段长度最高达1 kb的所有Illumina文库,无需考虑文库类型和GC含量。
KAPA SYBR FAST是一种抗体介导的热启动 DNA聚合酶制剂。因此,KAPA文库定量试剂盒适用于自动化液体处理系统,进行高通量样品定量。
产品应用
Illumina平台的KAPA文库定量试剂盒旨在为准备Illumina测序的文库进行准确和可重复的定量。只要文库的浓度 > 0.0002 pM,且含有与引物预混液(10x)中的引物互补的序列,无论哪种文库类型、文库构建方式和使用哪种Illumina测序设备,均可以使用该试剂盒进行定量。
该试剂盒可定量GC含量高和平均片段长度高达1 kb的文库。
除了NGS文库定量,该试剂盒还可用于检测Illumina文库构建过程中的工作场所的文库污染。
KAPA文库定量检测包括可以轻易实现自动化的重复移液步骤。强烈推荐将自动化液体处理系统用于高通量NGS处理流程。有关更多信息,请参阅原说明书KAPA文库定量技术指南。
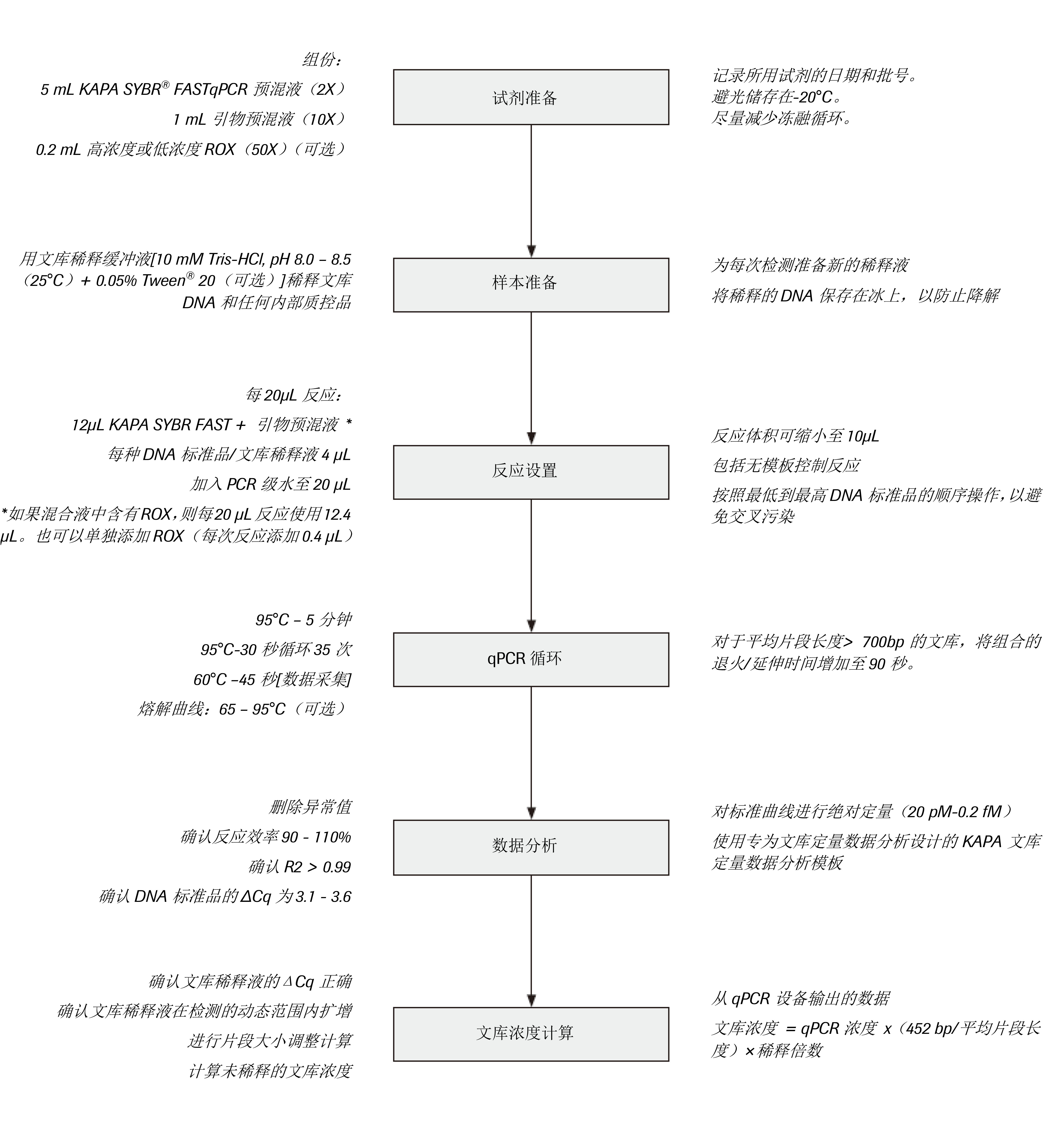
工作流程图
实验操作流程
-
-
-
- 试剂准备
1.1准备适量的DNA稀释缓冲液[10 mM Tris-HCl,pH 8.0-8.5(25 °C) + 0.05% Tween® 20(可选)]。该缓冲液可储存在室温或4 °C,并可重复使用。需始终在使用前将缓冲液平衡至室温。1.2需确保KAPA文库定量试剂盒的所有组分都完全解冻并彻底混合。1.3如果首次使用该试剂盒,将引物预混液(10x)(1 ml)加入KAPA SYBR® FASTqPCR预混液(2x)(5 ml)的瓶中。使用涡旋混合器彻底混合。
注意:如果您使用的是通用qPCR预混液试剂盒,且仅使用高浓度ROX染料或低浓度ROX染料,则在首次打开试剂盒时,可将适合的ROX溶液(50x)(0.2 ml)和引物添加到qPCR预混液中。应相应地调整每次反应所用的该混合液的体积(每20 μl反应使用12.4 μl或每10 μl反应使用6.2 μl)。1.4记录所有试剂的批号,以及引物(和ROX)被添加到qPCR预混液的日期。含有引物(和ROX)的KAPA SYBR FASTqPCR预混液经过30次冻融循环仍能保持稳定,在不使用时应避光保存在-20 °C下。如果混合液在反应设置的制备或随后使用过程中并未被微生物和/或核酸酶污染,那么可以在4 °C的暗处储存 ≤ 1周。 - 样本准备
2.1准备合适的文库稀释液(使用DNA稀释缓冲液)。根据文库的预期浓度,可采取1:1,000-1:100,000的稀释度。建议对每个文库额外进行至少一次2倍稀释。2.2
准备所需的内部质控稀释液(表1)。
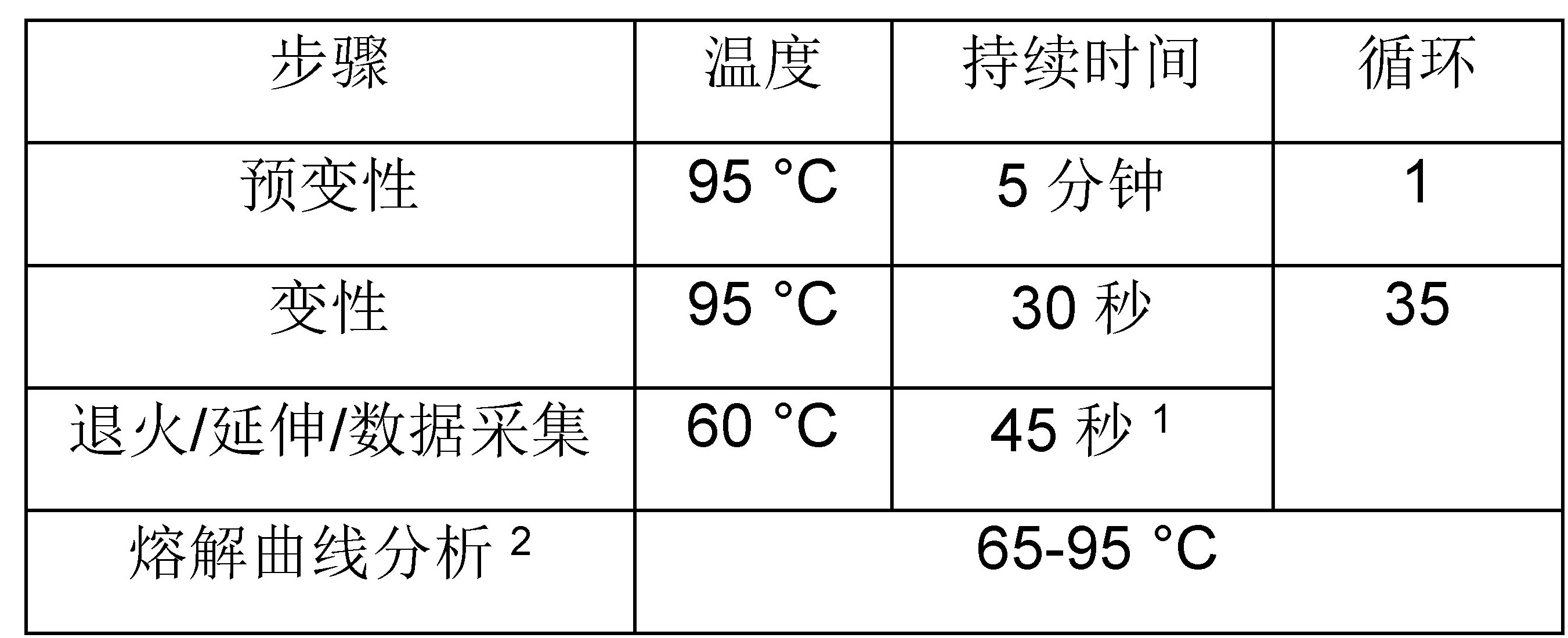
- 反应设置和循环
3.1确定以下每个反应的适当重复次数所需进行的反应总数:
● 6份DNA标准品
● 待测文库的每个稀释度
● 内部质控的每个稀释度
● 无模板质控品(NTCs)。3.23.3混合并短暂离心试剂预混液。3.4将适量预混液分配到每个PCR管或孔中。3.5向所有NTC孔/管中加入4 μl PCR级水。3.6按照从最低浓度(标准品6)到最高浓度(标准品1)的顺序,将4 μl每种DNA标准品分配到适当的孔/管中。3.7吸取4 μl每种稀释的文库和内部质控品进行分析。3.8盖上管或密封PCR板,并转移到qPCR设备上。 - 数据分析
注:在工作示例(步骤5)中提供了文库定量数据分析的工作示例。4.14.2检查扣除背景的(标准化的)扩增曲线和重复数据点(DNA标准品、文库和质控品)的Cq值,并排除明显的异常值。重复数据点应相差 ≤ 0.2个循环。如果数据集包含许多异常值,则结果不太可靠。进行重复检测,特别注重提高移液的准确性。4.3排除超出检测动态范围的所有文库稀释液,即去掉低于标准品1或高于标准6的平均Cq值。如果文库的所有稀释度都超出标准曲线,则重新定量一个更合适的文库稀释度。4.4使用设备软件生成标准曲线。也可以使用KAPA文库定量数据分析模板手动生成标准曲线。4.5查看标准曲线以确保满足以下条件:
● DNA标准品之间的平均ΔCq值在3.1-3.6的范围内。
● 计算的反应扩增效率在90-110%的范围内(即,PCR产物每循环增加1.8-2.2倍,标准曲线的斜率在-3.1和-3.6之间)。
● R2 ≥ 0.99。如果标准曲线不符合这些标准,则计算的文库浓度将不可靠,必须重复测定。4.6大多数qPCR软件会使用针对标准曲线的绝对定量来计算经稀释的文库和质控的浓度。但是,我们建议将qPCR数据导出到KAPA文库定量数据分析模板,以执行以下计算从而确定未稀释的文库浓度:
● 使用标准曲线将每个文库和内部质控品的每个稀释度的平均Cq值转换为平均浓度(以pM计)。
● 通过计算的平均浓度乘以以下因子,计算测定的每个文库和质控的每个稀释液的平均片段大小调整浓度(以pM计):
有关片段大小调整计算的更多信息,请参阅数据分析和解读(原说明书第9页)。
● 根据每个文库或质控品的每种稀释度乘以适当稀释倍数,再根据文库或质控品的长度,计算来自每种测定文库或质控品的原始浓度。4.7检查最终计算的浓度,并确定用于下游处理的每个样本的工作浓度(例如,用于靶向富集或成簇扩增的收集)。 - 工作示例
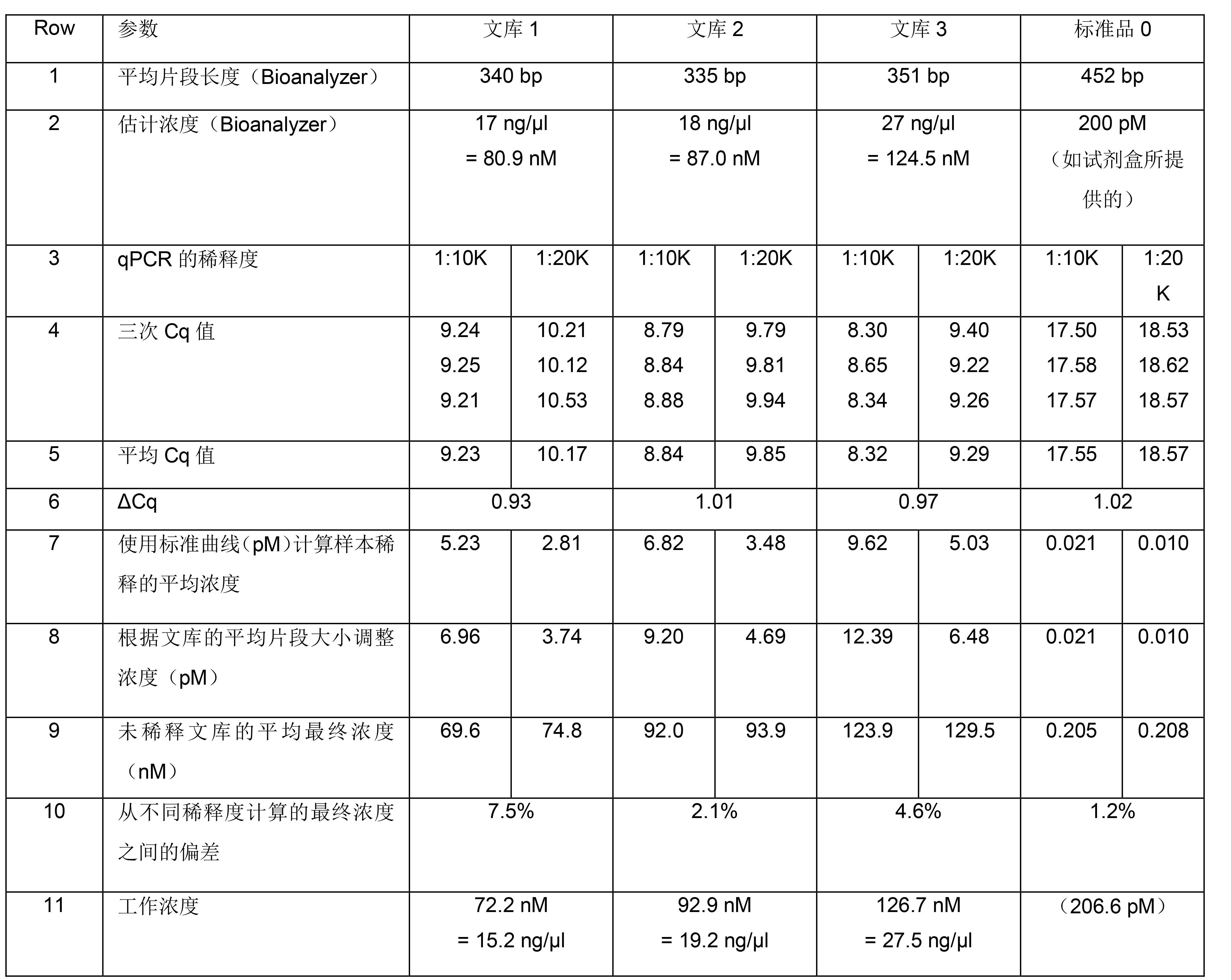
使用KAPA LTP文库构建试剂盒从250 ng经Covaris片段化的人类基因组DNA出发,制备3个带Index的DNA文库,适用于Illumina HiSeq 2500的2 × 100 bp配对末端全基因组测序。对接头连接的文库进行片段选择(250-450 bp)并扩增(6个循环)。使用Agilent Bioanalyzer高灵敏度DNA检测试剂盒分析扩增的文库,以确定每个文库的平均片段大小和大致浓度(表2,第1行和第2行)。为每个文库制备初步的1:10,000稀释液和另外一份2倍(即1:20,000)稀释液。作为流程质控,还制备了1:10,000和1:20,000稀释的KAPA文库定量内部质控品(Illumina DNA标准品0,200 pM)。使用KAPA文库定量试剂盒检测样本。所有DNA标准品和文库稀释液均需进行3次重复检测,对NTCs也进行3次重复检测。6种DNA标准品和NTCs的三次重复检测和平均Cq值将在下表中给出。
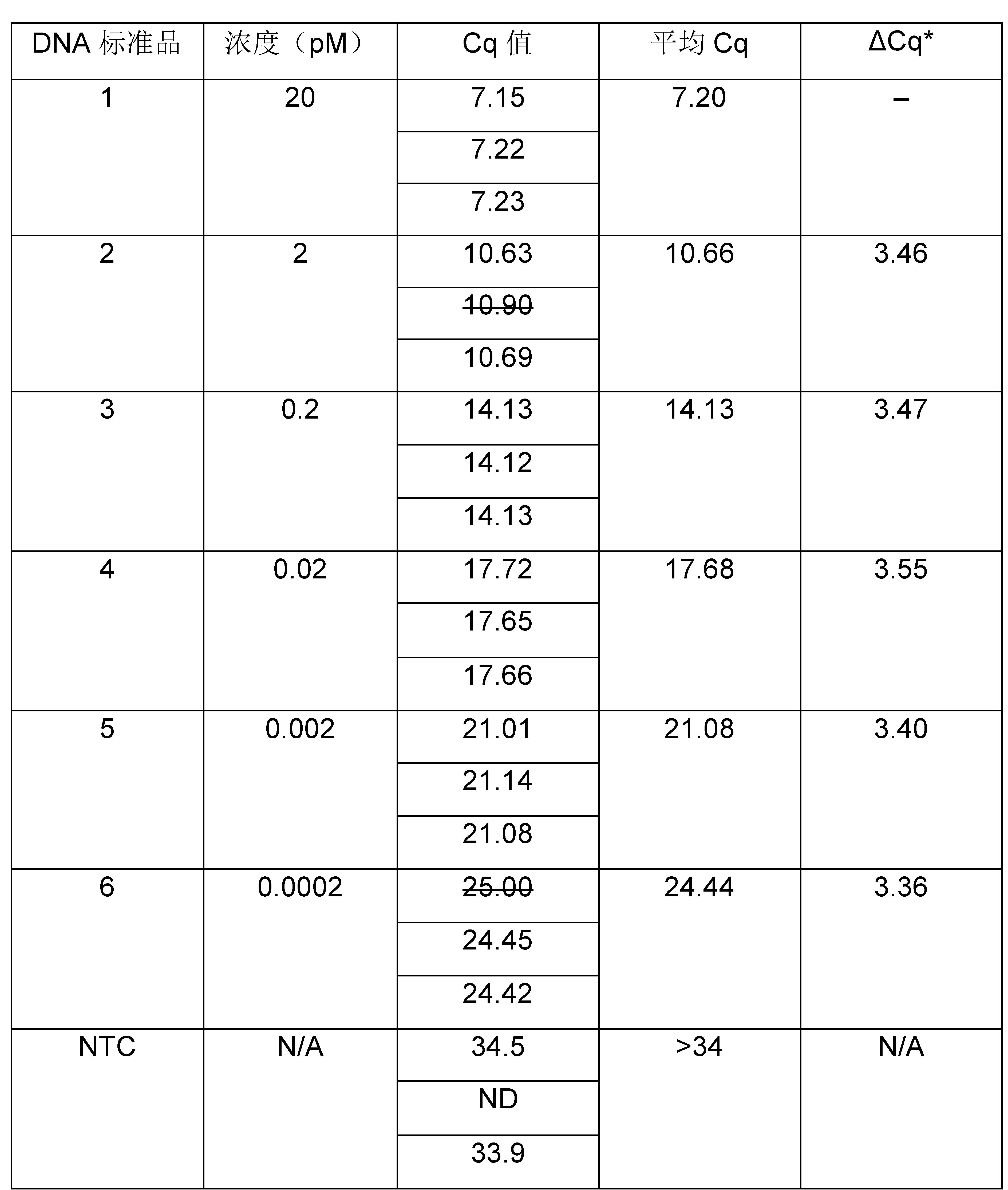
*对于10倍系列稀释的模板,应为3.1-3.6。排除DNA标准品2和6的异常值(> 0.2 Ct差异)后,生成标准曲线(图1)。标准曲线的质量控制指标如下:
● 所有相邻两个DNA标准品的ΔCq均在3.1-3.6的特定范围内。
● 95%的扩增效率在90-110%的特定范围内。
● R2值为0.9999符合 ≥ 0.99的技术参数。
图1.使用Illumina测序平台的KAPA文库定量试剂盒生成标准曲线表2第4行给出了3个文库和内部质控品的每个稀释度的三份Cq值。在计算每份样本的平均Cq值(第5行)之前,排除文库1的1:20,000稀释度和文库3的1:10,000稀释度的异常值(> 0.2 Ct差异)。
为了指示所计算浓度的可靠性,需要计算每个文库和内部质控的两个稀释度的ΔCq。对于一份样本的连续2倍稀释,预期ΔCq为1.0,并且介于0.9和1.1之间的值是可接受的。这表明,由两种或更多种稀释度计算的文库浓度可能相差 < 10%。如果样本连续稀释的ΔCq值落在可接受的范围之外,则表明由于液体处理不佳和/或样品污染,定量结果可能不可靠。
为了计算每个文库的工作浓度,需确定两种不同稀释度之间的差异。由于所有文库的计算浓度均存在< 10%的差异,故选择两种稀释液的平均值作为工作浓度。如果两个值相差> 10%,则工作浓度应基于以下之一:
● 从最终计算浓度值差异< 10%的稀释液得出的平均值。
● 每个文库的最低稀释度的最终计算浓度应该是可靠的。接下来,使用标准曲线将每个文库/质控稀释液的平均Cq值转换为浓度(以pM计)(表2,第7行)。随后根据每个文库的平均片段大小计算文库浓度(如实验操作流程中所述(步骤4.6);表2,第8行)。最后,根据每个测定的稀释液浓度,计算每个未稀释文库和质控品的平均终浓度(以nM计)(表2,第9行)。内部质控品的计算浓度(分别为1:10,000和1:20,000稀释度为205.4 pM和207.8 pM)在给定值 ± 10%范围内,表明在样本稀释或检测设置和执行过程中没有发生严重错误。
表2. 用于Illumina测序的基于qPCR的文库定量的工作示例
- 试剂准备
-
-
使用过本产品的部分文献
-
-
- Carpenter, M. L., Buenrostro, J. D., Valdiosera, C., Schroeder, H., Allentoft, M. E., Sikora, M., Rasmussen, M., Gravel, S., Guillen, S., Nekhrizov, G., Leshtakov, K., Dimitrova, D., Theodossiev, N., Pettener, D., Luiselli, D., Sandoval, K., Moreno-Estrada, A., Li, Y., Wang, J., Gilbert, M. T., Willerslev, E., Greenleaf, W. J. and Bustamante, C. D. (2013). Pulling out the 1%: whole-genome capture for the targeted enrichment of ancient DNA sequencing libraries. Am J Hum Genet 93(5): 852-864.
- Colman, R. E., Schupp, J. M., Hicks, N. D., Smith, D. E., Buchhagen, J. L., Valafar, F., Crudu, V., Romancenco, E., Noroc, E., Jackson, L., Catanzaro, D. G., Rodwell, T. C., Catanzaro, A., Keim, P. and Engelthaler, D. M. (2015). Detection of low-level mixed-population drug resistance in mycobacterium tuberculosis using high fidelity amplicon sequencing. PLoS One10(5): e0126626.
- Iha, C., Grassa, C. J., Lyra, G. M., Davis, C. C., Verbruggen, H. and Oliveira, M. C. (2018). Organellar genomics: a useful tool to study evolutionary relationships and molecular evolution in Gracilariaceae (Rhodophyta). J Phycol 54(6): 775-787.
- Kasparovska, J., Pecinkova, M., Dadakova, K., Krizova, L., Hadrova, S., Lexa, M., Lochman, J. and Kasparovsky, T. (2016). Effects of isoflavone-enriched feed on the rumen microbiota in dairy cows. PLoS One 11(4): e0154642.
- Symons, J., Chopra, A., Malatinkova, E., De Spiegelaere, W., Leary, S., Cooper, D., Abana, C. O., Rhodes, A., Rezaei, S. D., Vandekerckhove, L., Mallal, S., Lewin, S. R. and Cameron, P. U.(2017). HIV integration sites in latently infected cell lines: evidence of ongoing replication. Retrovirology 14(1): 2.
-