适用说明:本说明书适用于货号为KK8600,KK8601及KK8602的产品。
友情提示:
1)本文档中的页码,附录均指原说明书中的页码及附录;
2)本文档包含原说明书中的产品描述,产品应用及实验操作流程等主要信息,欲了解关于本产品详细说明及信息,建议参考原说明书 (点击面板上“下载PDF”可下载)。
产品描述
用于酶促片段化的 KAPA Frag 试剂盒可对多种不同类型和起始量(1 ng – 1 μg)的双链 DNA(dsDNA) 进行稳定可重复的酶促片段化,还可以整合入任何需要起始投入片段化 dsDNA 的 NGS 文库构建工作流程中。
该工作流程可实现自动化,与机械性剪切不同,该流程不需要任何专门的设备或耗材。片段化的程度(DNA 片段的主峰大小和大小分布)是由片段化时间和温度来控制的。最佳的片段化参数在某种程度上取决于起始 DNA 的数量和性质,但 KAPA Frag 系统对 DNA 起始量和质量的灵敏度要低很多,因此比其他酶促片段化技术(包括标记)的可重复性更高。
使用 KAPA Frag 系统进行片段化的 DNA 生成的文库,在功能上等同于通过 Covaris 片段化的DNA 制备的文库。
产品应用
KAPA Frag 试剂盒非常适合于对用于NGS 文库构建的 dsDNA 进行低通量和高通量的片段化。它兼容高复杂度的基因组DNA,也兼容低复杂度样本,如小病毒基因组、质粒、cDNA 和长扩增子,以及低质量DNA,如 FFPE 样本片段化DNA 可用于不同的NGS 应用,包括:
● 全基因组鸟枪法测序
● 全外显子组测序或靶向测序, 使用 Roche SeqCap EZ、Agilent SureSelect、Illumina TruSeq、IDT xGen Lockdown™探针或其他杂交捕获系统
● 长扩增子的测序
● 选择的 RNA 测序应用。
KAPA DNA HyperPlus 文库构建试剂盒将结合KAPA Frag 和 KAPA HyperPrep 化学方法,在同一个试管中对您的样本进行片段化/文库构建的操作,可为您提 供 更 高 的 文 库 产 量 。 请 访 问www.sequencing.roche.com 获取更多信息。
片段化实验方法
-
酶促片段化如果DNA 样本含有 EDTA,则使用 KAPA 纯化磁珠进行基于 3X 磁珠的纯化,以在片段化之前去除EDTA。有关详细的DNA 纯化实验方法, 请参阅 KAPA 纯化磁珠(KAPA 纯化磁珠)技术数据表。
或 者 , 准 备 足 够 体 积 的 适 当 稀 释 的Conditioning 溶液(每个DNA 样本准备 5 μL, 再加上过量的部分)。有关稀释 Conditioning 溶液的指导,请参阅表 2(第 4 页)。
1.1参照如下方法,稀释用于文库构建的 dsDNA 量:
● 如果DNA 制剂不含 EDTA,则在 35 μL 的总体积中用 10 mM Tris-HCl(pH 8.0-8.5)进行稀释
● 如果DNA 制剂确实含有 EDTA,则在 30 μL 的总体积中,用含有 EDTA、且样本目前悬浮其中的缓冲液进行稀释。对每一个包含30 μL 含EDTA 的DNA 的反应,加入 5 μL 稀释的 Conditioning 溶液。1.2通过轻轻涡旋或上下移液进行混合1.3按以下顺序添加剩余组分,在冰上配制每一个片段化反应:

* 可以将 KAPA 片段化缓冲液和酶预先混合,并在反应体系配制之前保存在冰上请注意,酶的体积大于此反应中的缓冲液体积。1.4轻轻涡旋并瞬时离心沉降。将 PCR 板/管放回冰上。立即继续下一步。1.5在 PCR 仪中进行孵育,预冷却至 4 °C,并按下述方法进行程序设置。将盖子的温度设置为 ≤ 50 °C。

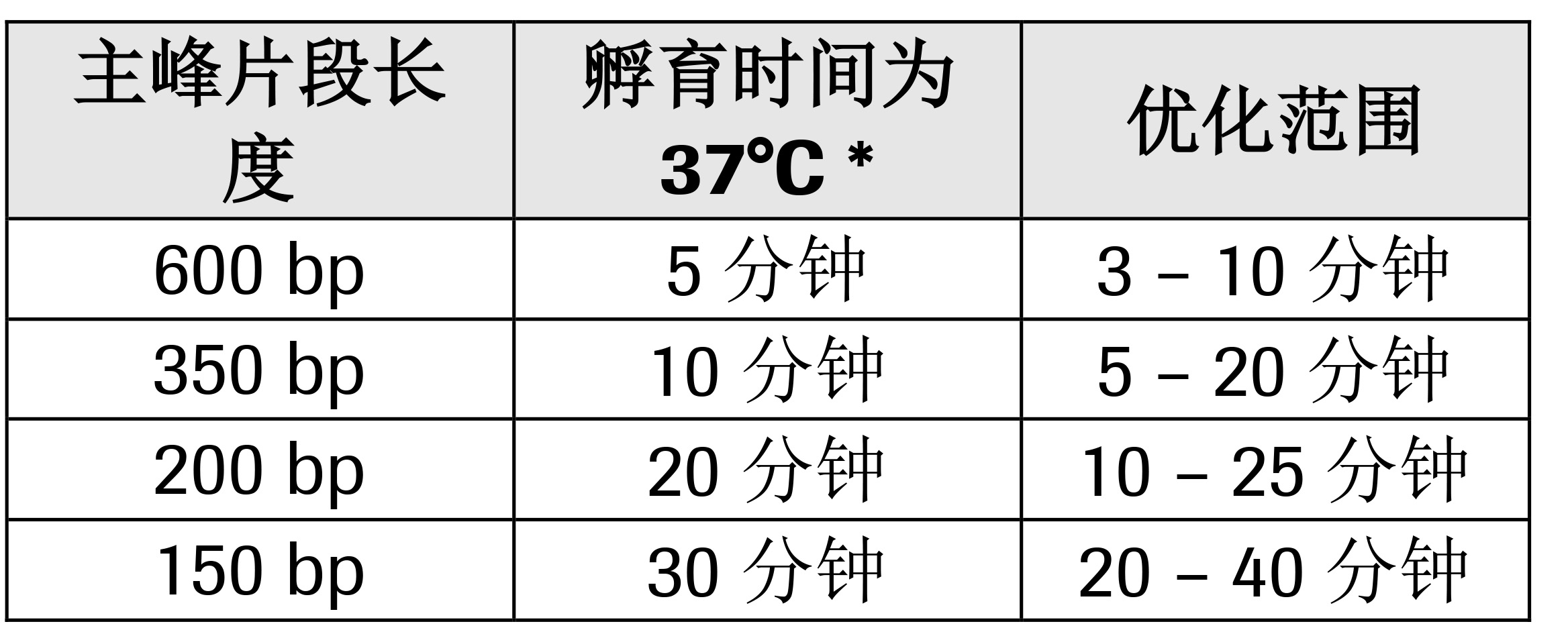
* 这些参数对于高质量基因组DNA 是一个不错的开始。有关如何优化片段化时间和温度的指南,参见反应优 化章节(第 5 页)。如果需要孵育时间的长度超过推荐范围,则样本可能会含有影响片段化效率的抑制剂。 建议在片段化之前进行基于磁珠的 DNA 纯化,而不是增加片段化的时间。1.6将反应转移至冰上,并立即添加 5 μL 终止液。轻轻涡旋,并直接进行片段化后纯化(步骤 2) 或片段化后片段选择(步骤 3)。 - 片段化后纯化
2.1结合以下方法,进行一次基于磁珠的纯化:

* 标准实验方法适用于 2X 的磁珠与 DNA 比率。有关更多详细信息以及有关如何修改纯化参数的指南,请参阅重要参数(第 3 – 4 页)。2.2通过涡旋和/或多次上下移液进行彻底混合。2.3将板/管在室温下孵育 5 – 15 分钟以将DNA 结合到磁珠上。2.4将板/管置于磁力架上以捕获磁珠。孵育直至液体澄清。2.5小心吸出并丢弃上清液。2.6将板/管置于磁力架上,加入 200 μL 80%乙醇。2.7在室温下将板/管于磁力架上孵育 ≥ 30 秒。2.8小心吸出并丢弃上清液。2.9将板/管置于磁力架上,加入 200 μL 80%乙醇。2.10在室温下将板/管于磁力架上孵育≥30 秒。2.11小心吸出并丢弃上清液。在不影响磁珠的情况下,尝试去除所有残留的乙醇。2.12在室温下将磁珠干燥 3-5 分钟,或直至所有乙醇挥发。
注意:过度干燥磁珠可能会导致产量降低。2.13从磁力架上取下板/管。2.14将磁珠重悬于适当体积的洗脱缓冲液(10 mM Tris-HCl,pH 8.0 – 8.5)中:
● 如果加入片段化的起始量在1 – 100 ng 的范围内,则重悬于 10 – 25 μL 的体积中。最小体积取决于所用磁力架的性质。
● 如果加入片段化的起始量 > 100 ng,则重悬于 30 – 55 μL 的体积中。2.15通过涡旋和/或上下移液进行彻底混合。2.16将板/管在室温下孵育 2 分钟以从磁珠上洗脱 DNA。2.17将板/管置于磁力架上以捕获磁珠。孵育直至液体澄清。2.18将澄清的上清液转移到新的 PCR 板/管中。将片段化的文库在 2 ℃至 8 ℃下储存 1-2 周,或在-15 °C 至-25 °C 储存。
- 片段化后双侧片段选择
为了获得片段化 DNA 的较窄片段分布,尤其是当片段化起始量 > 100 ng 时,和/或所需主峰片段长度超过 350 bp 时,在片段化后建议使用任何常用的片段选择技术(例如,此处介绍的双侧片段选择,或电泳法)。本章节中概述的双侧片段选择实验方法,是设计用于选择范围在 250 – 450 bp 的DNA 片段。为了获得更短或更长的片段,可以按照如下方式修改实验方法:
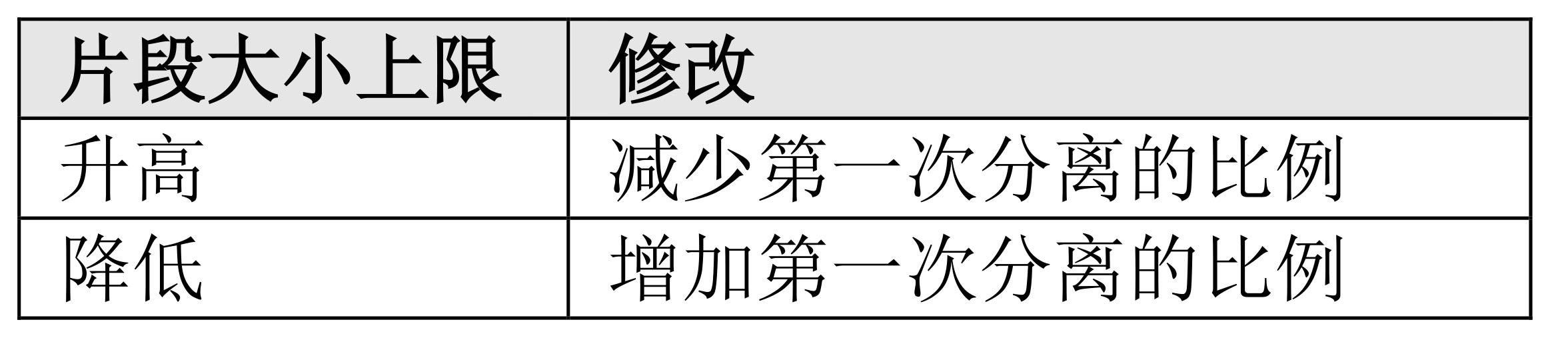
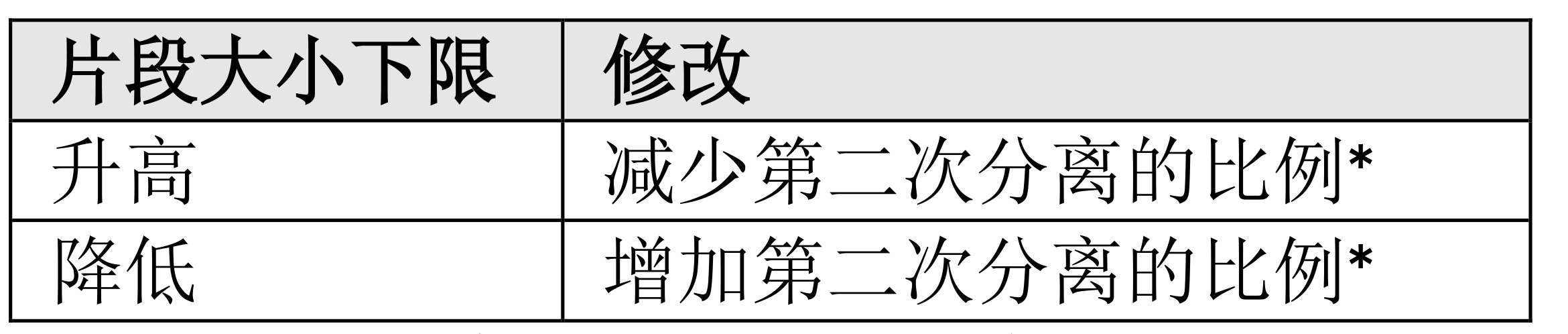
* 第二次片段分离应使用至少 0.2 个体积的 AMPure XP。请注意,第二次分离所需的 KAPA 纯化磁珠的体积是相对于片段选择过程开始时的 DNA 体积计算得来的,而不是第一次分离后转移的含 DNA 上清液的体积。如果第一次和第二次分离之间的差异小于~0.2 个体积,则 DNA 回收量显著降低。为了增加 DNA 回收量,可以使用> 0.2 体积的 KAPA 纯化磁珠用于第二次分离,但是注意这可能导致文库片段回收偏小和 /或片段大小分布更宽。
有关双侧片段选择的更多信息,请参阅 KAPA NGS 文 库 制 备 技 术 指 南 , 或 通 过 sequencing.roche.com/support 联系技术支持。
3.1结合下述方法,进行第一次(0.6X)片段大小分离(以去除大于~450 bp 的片段):
 3.2通过涡旋和/或多次上下移液进行彻底混合。3.3将板/管在室温下孵育 5 – 15 分钟,以将大于~450 bp 的 DNA 片段结合到磁珠上。3.4将板/管置于磁力架上以捕获磁珠。孵育直至液体澄清。3.5仔细地将含有小于~450 bp 的 DNA 片段的 85 μL 上清液移液到新的板/管中。
3.2通过涡旋和/或多次上下移液进行彻底混合。3.3将板/管在室温下孵育 5 – 15 分钟,以将大于~450 bp 的 DNA 片段结合到磁珠上。3.4将板/管置于磁力架上以捕获磁珠。孵育直至液体澄清。3.5仔细地将含有小于~450 bp 的 DNA 片段的 85 μL 上清液移液到新的板/管中。
重要的是,不要将磁珠与上清液一起转移。如果板/管中的磁珠结合了大于~450 bp 的DNA 片段,则丢弃这部分板/管。3.6结合下述方法进行第二次片段分离(0.8X,以保留 > 250 bp 的片段):
 3.7通过涡旋和/或多次上下移液进行彻底混合。3.8将板/管在室温下孵育 5 – 15 分钟,以将大于 ~ 250 bp 的 DNA 片段结合到磁珠上。3.9将板/管置于磁力架上以捕获磁珠。孵育直至液体澄清。3.10仔细地去除并丢弃含有小于~ 250 bp 的 DNA 片段的上清液。3.11将板/管置于磁力架上,加入 200 μL 80%乙醇。3.12在室温下将板/管于磁力架上孵育 ≥ 30 秒。3.13小心吸出并丢弃上清液。3.14将板/管置于磁力架上,加入 200 μL 80%乙醇。3.15在室温下将板/管于磁力架上孵育 ≥ 30 秒。3.16小心吸出并丢弃上清液。在不影响磁珠的情况下,尝试去除所有残留的乙醇。3.17在室温下将磁珠干燥 3-5 分钟,或直至所有乙醇挥发。
3.7通过涡旋和/或多次上下移液进行彻底混合。3.8将板/管在室温下孵育 5 – 15 分钟,以将大于 ~ 250 bp 的 DNA 片段结合到磁珠上。3.9将板/管置于磁力架上以捕获磁珠。孵育直至液体澄清。3.10仔细地去除并丢弃含有小于~ 250 bp 的 DNA 片段的上清液。3.11将板/管置于磁力架上,加入 200 μL 80%乙醇。3.12在室温下将板/管于磁力架上孵育 ≥ 30 秒。3.13小心吸出并丢弃上清液。3.14将板/管置于磁力架上,加入 200 μL 80%乙醇。3.15在室温下将板/管于磁力架上孵育 ≥ 30 秒。3.16小心吸出并丢弃上清液。在不影响磁珠的情况下,尝试去除所有残留的乙醇。3.17在室温下将磁珠干燥 3-5 分钟,或直至所有乙醇挥发。
注意:过度干燥磁珠可能会导致产量降低。3.18从磁力架上取下板/管。3.19将磁珠彻底重悬于所需体积的洗脱缓冲液(10 mM Tris-HCl,pH 8.0-8.5)。3.20将板/管在室温下孵育 2 分钟以从磁珠上洗脱DNA。3.21将板/管置于磁力架上以捕获磁珠。孵育直至液体澄清。3.22将进行了片段选择的DNA 转移到新的板/管中, 并将 DNA 在 2 °C 至 8 °C 下保存 1 至 2 周,或保存在-15 °C 至-25 °C。
